AlloSeq Tx17
AlloSeq Tx 17 to innowacyjne rozwiązanie NGS dla laboratoriów zajmujących się typowaniem antygenów HLA, które charakteryzuje się najszerszą paletą poddawanych analizie genów (17 loci). AlloSeq Tx17 wykorzystuje rekcję hybrydyzacji z sondami i wyłapywania (Hybrid Capture), co eliminuje ograniczenia tradycyjnych metod Long-Range PCR.
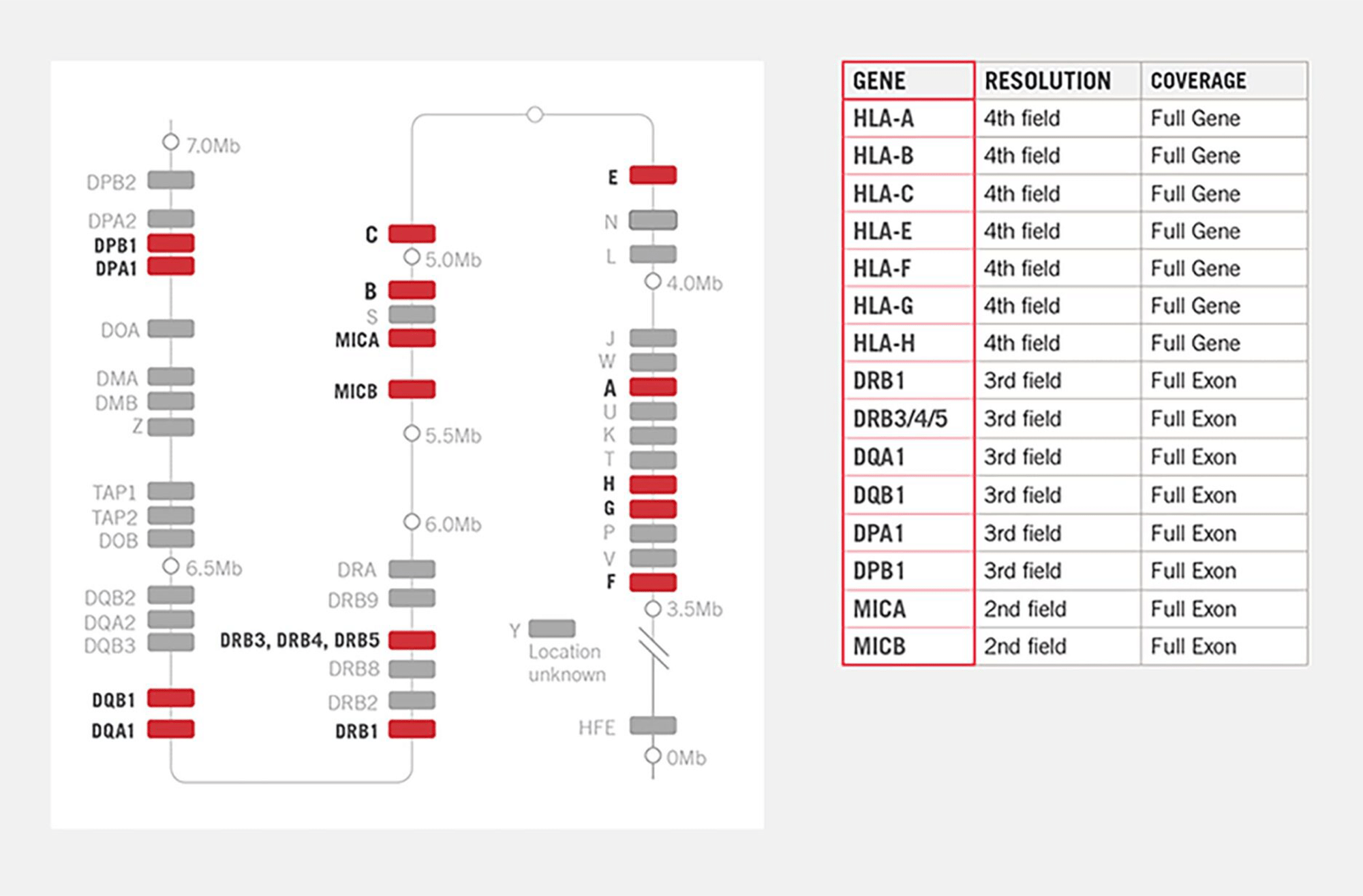
Analiza 17 loci z uwzględnieniem wszystkich egzonów
Sekwencjonowanie 17 loci uwzględnionych w panelu umożliwia najwyższy poziom dopasowania dawcy i biorcy. Producent zapowiada w niedalekiej przyszłości rozszerzenie listy genów, bez negatywnego efektu na zoptymalizowaną preparatykę oraz szybką analizę wyników w programie AlloSeq Assign.
Kilkuetapowy zoptymalizowany czasowo protokół
Protokół AlloSeq Tx17 pracy składa się z 4 etapów: enzymatycznej fragmentacji DNA przy użyciu transpozazy znajdującej się na kulkach, zapewniając równomierne cięcie gDNA, poprzedzonego pulowaniem prób wzbogacania (enrichment), sekwencjonowania bibliotek oraz analizy danych przy użyciu dedykowanego oprogramowania. Czas pracy manualnej to ok. 4 godziny.
Gwarancja precyzyjnej analizy wszystkich sekwencji docelowych
Zastosowanie technologii Hybrid Capture eliminuje ryzyko nierównego pokrycia sekwencji docelowych oraz zjawisko „drop-out” określonych fragmentów genów.
AlloSeq Assign - szybki import danych i zoptymalizowana analiza
Wykorzystanie oprogramowania AlloSeq Assign dedykowanego dla bibliotek AlloSeqTx gwarantuje przygotowanie raportu z wyników sekwencjonowania (pula 24 prób) w czasie do 30 minut.
Specyfikacja ogólna
|
Czas preparatyki |
8 h |
|
Czas pracy manualnej |
4 h |
|
Ilość materiału wejściowego |
> 50 ng |
|
Liczba reakcji |
24 lub 96 |
|
Rodzaj materiału wejściowego |
Krew |
|
Kompatybilność z systemami |
iSeq100, MiniSeq, MiSeq, MiSeqDx (tryb naukowy) |
|
Rodzaj kwasu nukleinowego |
DNA |
|
Gatunek |
Człowiek |
|
Metodyka |
Wzbogacanie próbek (enrichment) |
|
Technologia |
Sekwencjonowanie |